아카이브 논문
2025.10.20
링크드인에서 우연히 본 논문인데 biology 분야에서 생성모델을 어떻게 활용하는지 궁금했던터라 한번 살펴보기로 결심했다.
요약을 먼저 하자면,
이 논문에서는 세포, 핵 segmentation을 위해 병리 이미지를 생성하는 MSDM이라는 모델을 제안하고 있다.
결과적으로 segmentation model이 다양한 병리 이미지를 잘 분할하지 못하는 문제를 해결하고 싶었던 것 같은데, 이 논문에서는 segmentation model 자체의 모델 (구조) 개선이 아니라, 모델은 기존에서 잘 사용되는 모델을 그대로 사용하되, 성능을 개선하는 보조적인 방법으로써 데이터 증강을 시도했고, MSDM을 통해 합성한 병리 image-mask 쌍을 학습 데이터에 추가하여 데이터 증강을 함으로써 segmentation 모델의 성능 개선효과가 있는지를 검증하고자한 것 같다.
MSDM 구조를 살펴보고 -> MSDM을 통해 생성되는 이미지가 어떤지 살펴보고 -> 실제 segmentation model의 input, 즉 학습 데이터에 추가가 되었을 때 segmentation model의 성능이 어떻게 변했는지를 차례로 살펴보면 될 듯하다.
용어 정의
Segmentation model
- 병리학 이미지를 구성하는 각 세포, 핵 같은 미세 구조물을 픽셀 단위로 자동 구분해주는 딥러닝 기반 모델
Synthetic data
- 실제 수집한 데이터가 아니라, 인공적으로 만든 가짜 데이터
SDM (Semantic conditioned Diffusion Model)
- 기존 디퓨전 모델에 segment mask와 같은 semantic 정보를 추가해서 특정 구조, 형태에 맞춰 이미지를 생성할 수 있도록 한 모델
HV map
- 각 인스턴스(세포/핵)의 경계로부터 수평, 수직거리 맵
- 서로 붙어있는 세포의 인스턴스 구분에 효과적
Columnar cell
- 원통형, 길쭉한 세포
- 초기 학습 데이터셋에서 희소함
SPADE(spatially-adaptive normalization)
- 공간적 위치별로 다른 scale, bias 파라미터를 학습하여 semantic 단위(의미 단위)별로, semantic 정보를 공간적으로 보존하면서 정규화
Abstract
1. Introduction
높은 퀄리티의 데이터셋은, labor-intensive하고, expert annotation을 필요로 한다. 하지만 이러한 높은 퀄리티의 데이터셋은 병리학 분야의 딥러닝에 있어서 꼭 필요하다. 특히 rare한 경우 높은 퀄리티의 데이터셋이 부족한 경우가 많은데, 이 문제를 해결하기 위해 합성 데이터(Synthetic data)가 등장했다.
본 논문에서는, MSDM(Multimodal Semantic Diffusion Model)을 제안하는데, 이 모델은 합성 면역조직화학 이미지(synthetic IHC images)를 생성하는 모델로, 이 모델의 강한 conditioning 능력은 드물게 나타나는 형태학적 패턴을 잘 잡아내지 못했던 기존 segmentation model의 한계를 극복하게 해준다.
MSDM의 목적
- 기존의 세포, 핵 annotation을 re-use해서 사실적이고, 픽셀 단위로 정밀한 image-mask pair를 생성하는 것
- 드물게 나타나는 형태학적 패턴(low prevalence morphologies), 즉 columnar cells에서 segmentation model의 성능을 향상시키는 것 => MSDM으로 생성한 columnar cells 이미지를 추가함으로써 segmentation model의 성능을 향상
2. Background
Synthetic data를 생성하는 방법
1. image-to-image translation(I2I)
- Pix2Pix: CNN, adversarial training - transfer images btw domains
2. DDPM(Denoising Diffusion Probabilistic Model)
- ex) H&E images, rare cancer datasets, gigapixel pathology images 생성
3. SDM(Semantically conditioned Diffusion Model)
- 특히 의료 이미지 생성에 많이 쓰임
- ex) brain MRI, H&E images
- U-Net기반인 건 디퓨전과 동일한데, 디코더에다가 semantic label maps를 추가한다는 게 차이점? 이때 SPADE(spatially-adaptive normalization)을 통해 추가
4. Text-conditioned diffusion model
- 자연어 프롬프트 활용
SDM을 computational pathology에서 활용시의 한계
: 병리 이미지는 수많은 작고 서로 붙은 세포들로 이루어져 있어서, 각 세포를 하나하나 구분하기 위해서는
기존의 “의미 정보(semantic)”만 사용하는 방법보다 공간 정보를 훨씬 더 정밀하게 활용하는 접근(fine-grained spatial conditioning) 이 필요하다.
=> 1. semantic conditioning & 2. text conditioning 을 통합하는 multimodal 접근을 하고자 하였다.
3. Methods

SDM의 기본 구조 + 16개의 채널(2개의 HV maps channels, 3개의 mean RGB values channels, 3개의 std RGB channels; 즉, 각 세포, 핵 당 8 채널이므로 합쳐서 16개)
cf. HV maps: horizontal and vertical maps that are calculated as the horizontal/vertical distances to the boundaries of each instance
SPADE noramlization 통해서 이 16개 channel mask guides the diffusion process
- SPADE는 spatially adaptive normalization으로, 픽셀 단위로 의미적 마스크 정보를 반영함.
- diffusion model의 U-Net decoder에서 이 채널들이 사용됨.
text conditioning 진행: BERT-tiny
=> 이미지 + 텍스트 multi-head cross-attention
multi-head cross-attention
- 이미지 feature(noisy image의 latent feature) + 텍스트 임베딩 fusion
- Q는 이미지 feature에서, K, V는 텍스트 feature에서 추출
- 모델이 공간적 위치별로 텍스트적 문맥 정보를 결합할 수 있게 함
- 8개의 attention head 병렬로 사용
latent space features
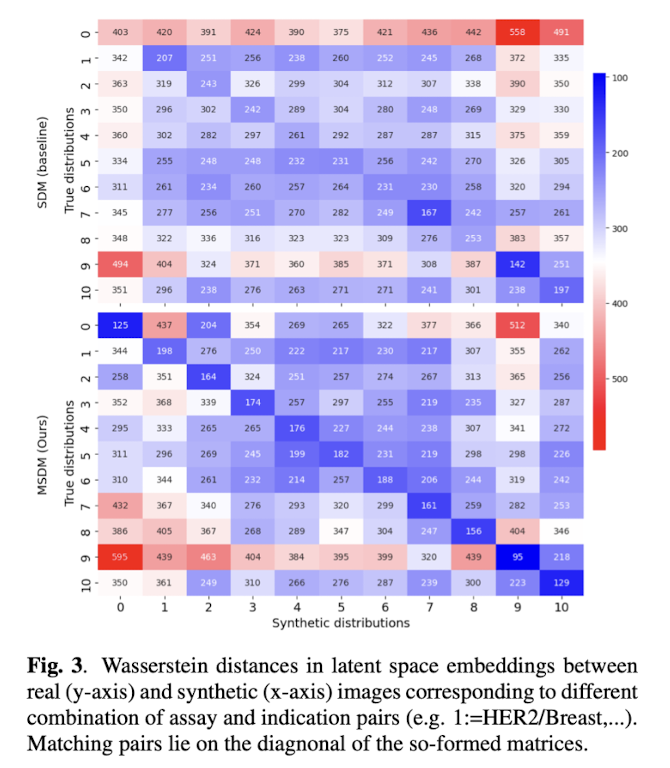
- 생성된 real 및 합성(synthetic) 이미지의 latent feature를 UMAP으로 시각화
- H-Optimus-0 foundation model을 사용하여 임베딩한 후, Wasserstein distance를 계산하여 실제 데이터 분포와의 차이를 정량적으로 평가
4. Experiments
4.1. Model Training
- Optimizer: Adam, loss = MSE, scheduler = cosine noise schedule.
- Epochs: 20,000
- Batch: 5 (with 5 gradient accumulation → effective batch = 25)
- Scheduler: DDIM (faster inference, 25× speedup vs DDPM).
- Validation: same across all experiments.
4.2. Datasets
- 총 566 IHC-stained slides (HER2 membrane marker, 20x) 사용.
- 이 중 612개 Field-of-View (FOV, 512×512 px)를 수집.
- 병리학자들이 세포 및 핵 윤곽을 라벨링함
- train set: 위 데이터 대부분
- validation set: 독립된 36개 IHC 슬라이드(30개 FOVs, 동일 20x 배율)
Annotation 수:
- 153,873 annotated cell outlines
- 110,834 annotated nuclei outlines
테스트 세트:
- Columnar cells 포함 세트
- 다양한 indication + cell 혼합 세트 (HER2/PI/P2/P3 등)
4.3. Dataset Enhancement with Diffusion Model
- columnar cells 중심으로 데이터 증강.
- 세포 이심률(eccentricity) 계산하여 원형성 평가
- 평균 eccentricity ≥ 0.85 인 120개 FOV을 선택.
- 각 FOV의 color/metadata 조건(randomly selected) 조합으로 synthetic FOV 생성.
- 총 960 synthetic FOVs 생성하여 augmentation.
4.4. Evaluation
- 세 가지 실험 조건으로 segmentation 성능 비교:
- Training with annotated real data only (baseline)
- Real data + synthetic images (SDM)
- Real data + synthetic images (MSDM)
- 세포/핵 segmentation model: Cellpose2
- 평가지표:
- F1 score (cell/nuclei segmentation)
- ASSD (average symmetric surface distance)
- Dice score (nuclei)
- IPA (agreement among three pathologists)
- 통계적 유의성 검정: Wilcoxon signed-rank test (p < 0.05)
- latent embedding 비교를 위해 UMAP + Wasserstein distance 계산.
Results
- MSDM은 두 가지 주요 평가에서 SDM보다 우수함:
- Text-guided image generation
- BERT-tiny를 통한 textual condition 반영 (assay + indication)
- synthetic 이미지가 실제 분포와 잘 align됨
- Wasserstein distance 측정 결과, MSDM이 SDM 대비 평균적으로 낮음 → 실제 데이터 분포를 더 잘 재현
- matching: 158.75 ± 29.50
- non-matching: 303.46 ± 69.73
- Downstream segmentation performance
- Columnar cells test set 기준, MSDM이 모든 지표에서 가장 높은 점수를 기록
- Cytoplasm/Cell boundary/Nuclei segmentation 모두 개선
- Text-guided image generation

- MSDM은 특히 세포 경계(cytoplasm) 및 핵 분할 성능 향상에 기여.
- 실험적 현실성을 위해 합성 이미지 품질(형태학적 일관성)도 높게 평가됨.
Discussion
- SDM을 병리영상용으로 확장하여 MSDM을 제안.
- HV map, RGB color, BERT metadata를 통해 morphology + condition-aware generation 가능.
- 특히 low-prevalence morphology (희귀 형태)에서도 segmentation 향상을 달성.
- 합성 데이터는 실제 데이터와 높은 morphologic 유사성을 보이며,
annotation 비용 절감 효과가 있음 (960 FOV 생성 시 약 3840시간 절약 추정). - 다만, 여전히 초기 라벨 데이터가 필요하다는 한계 존재.
- 향후에는:
- 다양한 morphology 및 assay 확장,
- multimodal fusion 모델의 병리학적 데이터 희소성 완화,
- 일반화 가능성 강화 방향으로 발전 예정.
